
 Yuichiro Abe
Yuichiro Abe
パトコア技術チームの安倍と申します。普段はプリセールス寄りのお仕事をしています。
技術ブログができたということで、せっかくなので年末の休暇中に読んだ面白い論本について、(だいぶ時間が経ちましたが。。)記事にしたいと思います。分子構造の様々な表記方法についてまとめた総説になっており、大変勉強になりました。
Molecular representations in AI-driven drug discovery: a review and practical guide David et al., J Cheminform 12, 56 (2020). DOI: https://doi.org/10.1186/s13321-020-00460-5
本記事は私が調べた内容も含まれるため、もし誤りがあればご指摘下さい。
目次
低分子
分子グラフ
 Graph Theory - Wikipedia
Graph Theory - Wikipediaほとんどの分子の表記方法はグラフ理論に基づいています。グラフ理論におけるノードとエッジを原子と結合に模して、それぞれに原子番号(元素名)や結合価等の属性を付与することで、分子構造を表現できます。そして、分子をグラフで表現することにより、行列、配列、木構造といった処理が可能なデータ構造に変換できるのです。
行列表記
MOL, SDF
現在、分子構造を扱うために広く用いられているファイル形式の一つとして、MDL(現Biovia社1)のMOL形式があります。MOL形式では原子に関する情報、結合に関する情報をConnection Table(CTab)形式で記録します。原子座標、結合様式、ステレオなど様々な情報を付与できるため、拡張性の高い形式となっています。V2000とV3000のバージョンの違いがあります。
SDF形式はMOL形式にプロパティを追加できる拡張形式です。CTabの下にその分子のプロパティを以下の形式で自由に追加できます。
> <property> value
MOL, SDF以外にも反応式を扱うためのRXNなどCTabを使った異なるファイル形式があるのですが、論文中ではわかりやすく以下の模式図にまとめられていました。

線形表記
MOLやSDFは多くの情報を記録できる利点がありますが、CTabで構造を表現するとどうしてもデータ量が増えます。ストレージやメモリの使用量を抑える必要がある場合、線形表記を使うとよりコンパクトに表現できます。
IUPAC名
化合物の正式名称として、IUPACの定める命名規則に基づいた化合物名は文献や特許などの正式な書類に残すためには必要不可欠です。一方でその命名規則は煩雑で、慣用名との組み合わせ等の誤用が頻発していますので、IUPAC名からの構造の名寄せは一筋縄ではいきません。IUPAC Color Booksに命名規則がまとめられていますが、定期的に更新されるため、IUPACが推奨する命名規則が変更される可能性もあります。情報量が膨大なの全部を理解している人はいるのでしょうかね。。
SMILES, CXSMILES, SMARTS
化学構造を線形表記で表現する方法として、かつてWLNというものが開発されていました。しかしその規則は煩雑であるため、より直感的に表現する方法として1988年に開発されたものがSimplified Molecular Input Line Entry System (SMILES)です。Daylight社2によりメンテナンスが行われており、現在様々な場面で使われている人気の高い表記方法となっています。
水素を除く原子をアルファベットで用いて、分岐を()で囲い、環を数字により表す、といったルールがあります。簡単な構造であれば手入力でも作れます。
例:安息香酸 OC(=O)c1ccccc1
SMILES表記は開始する原子の位置をずらす等により様々なバリエーションが存在し得るため、表記方法を一意にするために正規化(canonicalization)が必要であり、正規化したSMILESはcanonical SMILESと呼ばれます。このcanonicalization algorithmをDaylight社は公開していないため、SMILESは作成ツール(MarvinやRDKitなど)により若干異なります。
SMILESだけでは表現しきれない情報を扱うためにChemAxon Extended SMILES (CXSMILES)という形式も開発されており、拡張型SMILESとして用いられています。
また、SMILESが単一の分子構造を表現する一方、複数の分子構造を表現する方法として、SMILES Arbitrary Target Specification (SMARTS)というものがあります。Daylight社によりルールが定められており、Substructure Searchなどの構造検索に利用されます。一見すると難しく見えますが、ANDやNOTといったロジックを導入することや、原子回りの環境を規定するRecursive SMARTSを実現できるため、使いこなすと強力な表現方法となります。弊社のCRAIS Checkerという化学法規制チェックサービスでは、SMARTSによるクエリ構造の作成が必要不可欠な要素となっています。
InChI
The IUPAC International Chemical Identifier (InChI)はnon-proprietaryな化学表記方法として2006年にNISTとIUPACで共同開発され、現在は非営利組織であるInChI Trust3が開発を引き継いでいます。InChI作成用のツールもオープンソースで提供しています。
フォーマットとしては、/によりレイヤーを分けて分子式・結合・水素・ステレオなどの情報を付与する形式になっています。InChIそのままでは容量が大きくなるため、ハッシュ化して短縮表記となったInChIキーが大規模ライブラリにおいて用いられています。
InChIは低分子用の形式ですが、高分子・錯体・混合物・反応を表記するInChIの拡張版を開発中のようです。
分子記述子
ここまで説明した表記方法は原子と結合を表現して、そこから構造を再現することを目的としていました。一方で、物性やトポロジーといった要素を併せて表現することが、構造活性相関(QSAR)や機械学習によるデータ分析で必要とされます。用途に応じて多種多様な分子記述子が使われており、構造と特徴となる値を数値データに変換します。
分子記述子の中でも構造のトポロジーを0と1にビット化したものがフィンガープリントと呼ばれます。指紋により人物を特定できる様に、分子フィンガープリントは化合物の構造を表します。フィンガープリントを用いることで高速にライブラリーをスクーリングできるため、構造検索を行う際に用いられています。ChemAxonでは512あるいは1024ビットにハッシュ化されたフィンガープリントをデフォルトの構造検索に用います。
登録番号
厳密に言うと分子構造の表記方法ではありませんが、化合物を特定するために番号体系が用いられることがあります。社内データベースの化合物IDやパブリックデータベースのID(PubChem CID等)が当てはまります。代表的な例はChemical Abstracts Service4によるCAS登録番号です。登録番号自体に意味は無く(一部バリデーション機能を持つものもある)、分類のために附番されます。
CAS登録番号になるとCASのサービスを超えて様々な用途に使用されており、名寄せの需要も高いです。一方で、CAS登録番号の利用には使用料がかかるようになったため、現在ルックアップを利用できるサービスはCASの公式ツールか、NIHのCACTUSなどに限られています。弊社も試薬データベースSMARTSなどにCAS番号を利用しているため、毎年結構な金額の使用料を支払っています。
反応
有機反応の表記方法は、近年オープンデータとして反応データを使えるようになったことや、逆合成解析の注目を受けて活発に研究が進められている分野です。フォーマットとしては、これまで紹介した分子の表記に加え、反応点の特定及びReactant, Product, Catalyst, Reagent などの役割の指定が必要となります。この分野に詳しくないので詳細は総説を見ていただきたいですが、以下のファイル形式がよく用いられます。
- RD, RXN
- Reaction SMILES, SMIRKS
- RInChI
マクロ・高分子
ケモインフォマティクスが化学を扱うのに対して、生命科学を扱う学問領域がバイオインフォマティクスです。データ形式の観点から言うと、前者が分子構造を研究対象としているのに対して、後者ではタンパク質や核酸などのバイオポリマーをシーケンス(配列)として扱います。一方、近年になってコンピュータの計算処理能力が向上したこと、またペプチド・核酸・抗体薬物複合体(ADC)が創薬における新たなモダリティとして注目されるようになったことから、分野間の垣根は下がっています。この様な背景により、両分野を繋ぐためのマクロ・高分子の表記方法が必要とされています。
ペプチド, 核酸, 抗体, グリカン
モノマー略号
アミノ酸や核酸は略号として表記できます(例:アラニン A, Ala アデニン A)。天然型だけであれば略号を使うだけで良いですが、多様な非天然型アミノ酸や人工核酸の場合は詳細な表記方法が必要ですし、同時にシーケンス上で表す必要があります。つまり、バイオポリマーを表記するためにはモノマーの分子構造とシーケンスを相互変換させる必要があります。
HELM
製薬企業のコンソーシアムであるPistoia Alliance5により開発が続けらている表記方法がHierarchical Editing Language for macromolecules (HELM)です。ペプチド・核酸・抗体・グリカンなど、新たな創薬モダリティを表現できるため、現在主流になりつつあります。オープンソースのツールキットも開発されています。

形式としては、名前が示す様に構造情報を「Complex polymer」→「Simple polymer」→「Monomer」→「Atom」という階層に分けて表記します。モノマーにユニークなIDを割り当てることによりシーケンス表記と分子構造の情報を両立させることができます。モノマーIDの代わりにSMILESで表記することも可能なようですが、新規モノマー構造を登録するための仕組み作りも行われています。monomer.orgというサイトにてAPIを叩くことでモノマーライブラリーを取得できるようになるようです。
HELMについては別記事で詳細をまとめたいです(きっと、近いうちに)。
タンパク質

PDB
タンパク質もペプチドと同じくアミノ酸により構成されていますが、アミノ酸残基がおよそ50以上をタンパク質と呼びます。このタンパク質のデータベースとして構造生物学分野にとって必須の情報源がProtein Data Bank (PDB)です。現在15万件以上のタンパク質の構造情報が収載されているようです。構造情報はPDB形式のファイルの中にアミノ酸残基の名称・高次構造・全ての原子座標・溶媒やリガンド等を含めて記録されています。
PLN
PDBファイルはタンパク質の構造情報を詳細に記録した分野標準のファイル形式ですが、データ量が大きくなります。そのため、タンパク質をアミノ酸残基の線形表記でシンプルに表現する Protein Line Notation (PLN) が2008年にBiochemfusion社6により開発されました。現在、PubChemデータベースなどにも用いられています。
ポリマー
近年マテリアルズ・インフォマティクスの分野が注目されおり、材料系研究でもAIや機械学習が用いられるようになりました。ポリマーの表記もChemDraw, Marvin, BIOVIA Drawなどの構造式エディタで表現できますが、より扱いやすい線形表記方法が開発されています。
BigSMILES
現在MITのグループにより開発が行われている表記方法が、SMILESを拡張したBigSMILESです。繰り返し部位をSMILESで表記することができ、エンドグループや重合様式など材料系ポリマーに必要な情報を表現できるようです。Canonicalizationは開発中ということですが、注目すべきプロジェクトだと思います。
余談ですが、ポリマーを表現するのに必要なPDIや添加剤、プロセス等の付随情報をjson形式でまとめたCitrinationというプロジェクトもあるようですね。大変興味深いです。
画像表記
Markush構造

分子の構造はMarvinSketchなどの構造式エディタで描画できますし、3Dの構造もAvogadroやPyMOLを使って作成できます。その構造を画像として論文や特許に表記するために、Markush構造と呼ばれる表記方法がよく使われます。母骨格にRグループを付け、複数の置換基をグループ化することにより、数多くの分子構造を一つの構造で表現できる表記方法です。特に化学系特許において重要な表記方法であり、数千、数万通りの新規物質の特許請求範囲を明示するために複雑なMarkush構造が用いられます。
Markush構造は便利ですが、画像から自動で構造データに変換できない、個別の構造へ分解(R分解)するために特別なツールが必要という欠点があります。この目的のため、ChemAxon社からはChemCuratorやMarkush Editorが提供されています。
まとめ
弊社では化合物登録システムを開発していますので、個人的な勉強のために表記方法についてまとめてみました。誰かのお役に立てれば幸いです。
最後にアラニンの表記をまとめてみました。
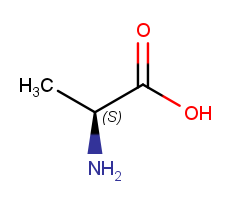
| 表記方法 | 表記 |
|---|---|
| 和名 | L-アラニン |
| Common Name | L-alanine |
| IUPAC Name | (2S)-2-aminopropanoic acid |
| SMILES | C[C@H](N)C(O)=O |
| InChI | InChI=1S/C3H7NO2/c1-2(4)3(5)6/h2H,4H2,1H3,(H,5,6)/t2-/m0/s1 |
| InChI Key | InChIKey=QNAYBMKLOCPYGJ-REOHCLBHSA-N |
| MOL(V2000) | l-alanine Mrv2102 02262111132D 6 5 0 0 1 0 999 V2000 0.8250 1.4289 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 1.2375 0.7145 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 2.0625 0.7145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 0.8250 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 1.2375 -0.7145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 0.0000 0.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 2 1 1 1 0 0 0 2 3 1 0 0 0 0 2 4 1 0 0 0 0 4 5 1 0 0 0 0 4 6 2 0 0 0 0 M END |
| PubChem CID | 5950 |
| HELM | PEPTIDE1{A}$$$$V2.0 |
| Three-letter symbol | Ala |
| PLN | H-{l}A-OH |
-
Dayligt https://www.daylight.com/↩
-
InChI Trust https://www.inchi-trust.org/↩
-
Pistoia Alliance https://www.pistoiaalliance.org/↩
-
Biochemfusion http://www.biochemfusion.com/↩
